友情链接:Datawhale-CV实践
GitHub项目地址:CV_Project
题目回顾
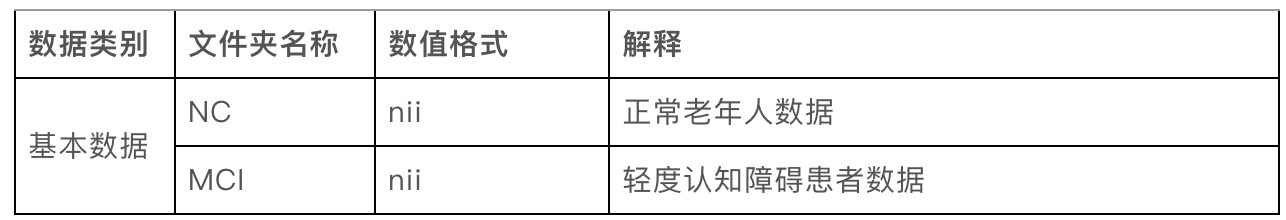
为研究基于脑PET图像的疾病预测,本次大赛提供了海量脑PET数据集作为脑PET图像检测数据库的训练样本,参赛者需根据提供的样本构建模型,对轻度认知障碍进行分析和预测。
脑PET图像检测数据库,记录了老年人受试志愿者的脑PET影像资料,其中包括确诊为轻度认知障碍(MCI)患者的脑部影像数据和健康人(NC)的脑部影像数据。
被试者按医学诊断分为两类:
NC:健康
MCI:轻度认知障碍
本次大赛所用脑PET图像检测数据库,图像格式为nii。
表1.大赛数据库
在此之前,对于cv仅限于知道
首先来认识一下nii格式文件
.nii文件
NII 格式是一种常用于医学影像数据的文件格式,它通常用于存储神经影像数据,如MRI(磁共振成像)和CT(计算机断层扫描)图像。NII 的全称是 Neuroimaging Informatics Technology Initiative,它是一种基于文件扩展名 “.nii” 或 “.nii.gz” 的文件格式。这些文件包含了图像数据和相关的元数据,如图像尺寸、像素分辨率、采集参数等。
以下是一些关键的信息,有助于了解 NII 文件格式:
图像数据: NII 文件包含了图像的原始像素数据。这些数据可以表示为多维数组,其中每个维度对应于图像的不同方向(例如 X、Y 和 Z 轴),以及可能的时间维度。
头部信息: NII 文件的开头部分包含了关于图像数据的元数据,如图像尺寸、像素分辨率、采集参数(如重复时间、TR)、图像类型(例如 T1 加权还是 T2 加权)等信息。这些元数据通常以一种被称为 “NIfTI-1 头部” 的结构进行组织。
文件扩展名: NII 文件可以具有 “.nii” 或 “.nii.gz” 的文件扩展名。”.nii” 文件是未经压缩的原始图像数据,而 “.nii.gz” 文件是经过 gzip 压缩的版本,占用更小的存储空间。
工具和库: 有许多开源的工具和库可以用来读取、处理和分析 NII 格式的文件。其中一些工具包括 FSL(FMRIB Software Library)、ANTs(Advanced Normalization Tools)和 MRIcron 等。
如果您需要处理或分析 NII 格式的文件,您可以使用这些工具来读取文件内容、进行图像处理、进行数据分析等。常见的操作包括图像配准、脑部分割、病变定位等。请注意,NII 文件在医学影像研究和临床诊断中发挥着重要作用,因此在处理这些文件时需要谨慎并遵循相关的法律和伦理规定。
也就是说,可以把nii文件理解为一张三维的照片,可以从三个方向进行切片
那么这也就引出了baseline
baseline的实现
Baseline,基于Logistic Regression
导入相关库
使用glob库来获取文件路径,numpy用于数值计算,pandas用于数据处理,nibabel用于加载和处理医学图像数据,OrthoSlicer3D用于图像可视化,Counter用于计数统计
import glob # 获取文件路径
import numpy as np
import pandas as pd
import nibabel as nib # 处理医学图像数据
from nibabel.viewers import OrthoSlicer3D # 图像可视化
from collections import Counter # 计数统计数据预处理
# 读取训练集文件路径
train_path = glob.glob('脑PET图像分析和疾病预测挑战赛公开数据/Train/*/*')
test_path = glob.glob('脑PET图像分析和疾病预测挑战赛公开数据/Test/*')
# 打乱训练集和测试集的顺序
np.random.shuffle(train_path)
np.random.shuffle(test_path)特征提取
定义了一个函数extract_feature,用于从脑PET图像中提取特征。
extract_feature函数从文件路径加载PET图像数据,并从中随机选择10个通道。然后,它计算了一系列统计特征,如非零像素数量、零像素数量、平均值、标准差等。最后,函数根据文件路径判断样本类别,并将提取到的特征和类别作为返回值。
def extract_feature(path):
# 加载PET图像数据
img = nib.load(path)
# 获取第一个通道的数据
img = img.dataobj[:, :, :, 0]
# 随机筛选其中的10个通道提取特征
random_img = img[:, :, np.random.choice(range(img.shape[2]), 10)]
# 对图片计算统计值
feat = [
(random_img != 0).sum(), # 非零像素的数量
(random_img == 0).sum(), # 零像素的数量
random_img.mean(), # 平均值
random_img.std(), # 标准差
len(np.where(random_img.mean(0))[0]), # 在列方向上平均值不为零的数量
len(np.where(random_img.mean(1))[0]), # 在行方向上平均值不为零的数量
random_img.mean(0).max(), # 列方向上的最大平均值
random_img.mean(1).max() # 行方向上的最大平均值
]
# 根据路径判断样本类别('NC'表示正常,'MCI'表示异常)
if 'NC' in path:
return feat + ['NC']
else:
return feat + ['MCI']模型训练
利用extract_feature函数提取训练集和测试集的特征,并使用逻辑回归模型对训练集进行训练。
在这里,我们通过循环将特征提取过程重复进行30次,这是为了增加训练样本的多样性。然后,我们使用逻辑回归模型LogisticRegression来训练数据。在训练完成后,模型已经学习到了从特征到类别的映射关系
# 对训练集进行30次特征提取,每次提取后的特征以及类别('NC'表示正常,'MCI'表示异常)被添加到train_feat列表中。
train_feat = []
for _ in range(30):
for path in train_path:
train_feat.append(extract_feature(path))
# 对测试集进行30次特征提取
test_feat = []
for _ in range(30):
for path in test_path:
test_feat.append(extract_feature(path))
# 使用训练集的特征作为输入,训练集的类别作为输出,对逻辑回归模型进行训练。
from sklearn.linear_model import LogisticRegression
m = LogisticRegression(max_iter=1000)
m.fit(
np.array(train_feat)[:, :-1].astype(np.float32), # 特征
np.array(train_feat)[:, -1] # 类别
)
print(test_path)['脑PET图像分析和疾病预测挑战赛公开数据/Test\\50.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\43.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\60.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\81.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\24.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\54.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\18.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\45.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\52.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\76.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\65.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\96.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\11.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\4.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\7.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\84.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\82.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\55.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\35.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\13.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\47.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\27.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\80.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\15.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\30.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\53.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\41.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\95.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\62.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\51.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\72.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\39.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\28.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\33.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\98.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\97.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\19.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\57.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\58.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\25.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\14.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\74.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\66.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\67.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\2.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\44.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\63.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\70.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\90.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\49.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\61.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\83.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\17.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\87.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\34.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\92.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\5.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\71.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\36.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\23.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\79.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\59.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\29.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\1.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\32.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\64.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\16.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\94.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\21.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\3.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\73.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\89.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\93.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\40.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\12.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\99.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\91.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\31.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\77.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\42.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\56.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\86.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\20.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\75.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\9.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\68.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\100.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\10.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\46.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\26.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\78.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\48.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\22.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\8.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\6.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\37.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\69.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\38.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\85.nii', '脑PET图像分析和疾病预测挑战赛公开数据/Test\\88.nii']
预测与结果输出
使用训练好的逻辑回归模型对测试集进行预测,并将预测结果进行投票,选出最多的类别作为该样本的最终预测类别。最后,将预测结果存储在CSV文件中并提交结果。
具体来说,使用了Counter来统计每个样本的30次预测结果中最多的类别,并将结果存储在test_pred_label列表中。然后,将样本ID和对应的预测类别存储在一个DataFrame中,并将其按照ID排序后保存为CSV文件,这样就得到了最终的结果提交文件。
# 对测试集进行预测并进行转置操作,使得每个样本有30次预测结果。
test_pred = m.predict(np.array(test_feat)[:, :-1].astype(np.float32))
test_pred = test_pred.reshape(30, -1).T
# 对每个样本的30次预测结果进行投票,选出最多的类别作为该样本的最终预测类别,存储在test_pred_label列表中。
test_pred_label = [Counter(x).most_common(1)[0][0] for x in test_pred]
# 生成提交结果的DataFrame,其中包括样本ID和预测类别。
submit = pd.DataFrame(
{
'uuid': [int(x.split('\\')[-1][:-4]) for x in test_path], # 提取测试集文件名中的ID
'label': test_pred_label # 预测的类别
}
)
# 按照ID对结果排序并保存为CSV文件
submit = submit.sort_values(by='uuid')
submit.to_csv('submit.csv', index=None)跑出来的分数是0.48485,不愧是Baseline!
在实现baseline中的一些困惑,还有debug
- 特征提取中随机选择10个通道进行提取,然后计算的统计特征都是人为规定,故存在偶然性,这一次可能抽样抽的好就成绩好,抽的运气差点那就成绩也难看
- 关于split,对
\\进行划分会更好一点
初次尝试CNN
认识CNN
卷积神经网络(Convolutional Neural Network,CNN)是一种在计算机视觉和图像处理领域广泛应用的深度学习模型。它专门用于处理具有网格结构数据的任务,例如图像和语音等。CNN 在图像分类、目标检测、图像生成等领域取得了显著的成功,因其在处理图像等数据时的特殊设计。
以下是 CNN 的一些重要概念和组成部分:
卷积层(Convolutional Layer):卷积是 CNN 的核心操作之一。它通过滑动一个小的窗口(卷积核或过滤器)在输入数据(如图像)上,对局部区域进行特征提取。卷积操作可以捕捉到不同位置的特征,从而实现平移不变性。多个卷积核可以提取不同的特征。
池化层(Pooling Layer):池化操作用于降低特征图的尺寸,减少计算量,并保留重要的特征。最大池化和平均池化是常见的池化操作,它们分别取区域内最大值或平均值作为输出。
激活函数(Activation Function):激活函数引入非线性性质,使得网络能够学习复杂的模式。常用的激活函数包括ReLU(Rectified Linear Unit)、Sigmoid 和 Tanh。
全连接层(Fully Connected Layer):在卷积和池化层之后,一般会连接若干全连接层,用于将提取的特征映射到最终的输出类别。全连接层的神经元与上一层的所有神经元相连。
批归一化(Batch Normalization):用于在训练过程中对每一层的输出进行标准化,以加速网络的训练并提高稳定性。
Dropout:在训练过程中,随机将某些神经元置零,有助于防止过拟合。
CNN 的典型架构通常由一系列的卷积层、池化层、激活函数、全连接层等组成,最后通过一个适当的激活函数得到分类结果。深度卷积网络的层数可以很深,例如,VGG、ResNet 和 Inception 等网络。
在计算机视觉领域,CNN 以其在图像特征提取方面的强大能力而闻名,通过多次堆叠卷积层和池化层,网络可以逐渐学习到越来越抽象和高级的特征表示,从而在图像分类、物体检测、分割等任务中取得卓越表现。
分析思路
这段代码是一个深度学习项目的实现,主要用于处理脑PET图像数据,进行分类任务(识别脑PET图像是否为正常状态NC或认知障碍状态MCI)。以下是代码的详细解释:
导入库:首先导入了一系列需要用到的Python库,包括处理数据和图像的库(如NumPy、Pandas、PIL、OpenCV、albumentations等),PyTorch库,以及其他辅助库。
设置随机种子和GPU设置:通过设置随机种子确保实验的可重现性,以及设置PyTorch在GPU上运行的一些参数。
定义文件路径:指定训练和测试数据的文件路径。
train_path是训练图像的路径,test_path是测试图像的路径。数据缓存定义:
DATA_CACHE是一个用于存储图像数据的缓存字典,以避免在每次访问时都加载图像数据。自定义数据集类 XunFeiDataset:这个类用于创建一个自定义的数据集,用于加载和处理图像数据。
__init__方法初始化数据集,接收图像路径和一个可选的图像变换(transforms)。__getitem__方法负责加载和处理数据。首先,它从缓存中加载图像数据,如果缓存中没有,就从NIfTI文件加载数据。然后,它从加载的图像中随机选择一些通道(层数),对图像进行变换(如果有的话),最后返回处理后的图像和标签。__len__方法返回数据集的长度。
数据加载器定义:创建训练、验证和测试数据加载器,用于加载批量的图像数据。这里使用了
torch.utils.data.DataLoader来实现。定义神经网络模型 XunFeiNet:这个类定义了一个基于预训练的ResNet-18模型的自定义模型。
- 在构造函数
__init__中,加载了预训练的ResNet-18模型,并对其进行修改,以适应数据。首先,修改了第一个卷积层的输入通道数,使其适应输入数据的通道数(这里是50通道)。然后,修改了全连接层的输出大小,以适应分类任务(2个类别:NC和MCI)。 - 在
forward方法中,将输入数据传递给修改后的ResNet-18模型,并返回模型输出。
- 在构造函数
模型、损失函数和优化器定义:创建了一个 XunFeiNet 模型的实例,将其放置在GPU上运行。定义交叉熵损失函数和AdamW优化器。
训练函数 train:定义了一个训练函数,用于在训练集上训练模型。
- 在训练循环中,迭代训练数据加载器中的每个批次。
- 将输入数据和目标标签移到GPU上。
- 通过模型前向传播获取预测输出,计算损失。
- 清零优化器的梯度,进行反向传播和参数更新。
- 打印每20个批次的损失。
- 返回平均训练损失。
验证函数 validate:定义了一个验证函数,用于在验证集上评估模型性能。
- 在验证循环中,迭代验证数据加载器中的每个批次。
- 将输入数据和目标标签移到GPU上。
- 通过模型前向传播获取预测输出,计算损失。
- 统计预测正确的数量,用于计算准确率。
- 返回验证集上的准确率。
训练循环:使用训练函数和验证函数进行训练。
- 在循环内,迭代指定次数(这里是40次)进行训练和验证。
- 调用训练函数进行模型训练,获取训练损失。
- 调用验证函数计算验证集上的准确率以及训练集上的准确率。
- 打印每轮训练和验证的结果。
预测函数 predict:定义了一个预测函数,用于在测试集上进行预测。
- 在预测循环中,迭代测试数据加载器中的每个批次。
- 将输入数据移到GPU上,通过模型前向传播获取预测输出。
- 将预测输出存储在一个列表中。
- 返回预测结果的NumPy数组。
测试集预测循环:使用预测函数进行测试集上的多次预测,并将结果累加。
- 迭代指定次数(这里是10次)进行测试集预测。
- 第一次预测时,将预测结果存储在
pred变量中。 - 从第二次预测开始,将后续预测结果累加到
pred变量中。
结果生成和提交:使用预测结果生成一个DataFrame,将结果存储为CSV文件。
- 将测试图像路径中的UUID解析为DataFrame的 ‘uuid’ 列。
- 将预测结果中的类别转换为 ‘NC’ 和 ‘MCI’,存储为 ‘label’ 列。
- 将DataFrame按UUID排序。
- 将DataFrame保存为名为 ‘submit2.csv’ 的CSV文件,以便进行结果提交。
这段代码通过自定义数据集类、模型定义、训练和验证循环等组件,实现了对脑PET图像
进行分类的深度学习流程,并最终生成一个用于提交的CSV文件,其中包含了对测试图像的分类结果。
针对源码的修改
num_workers设置为0,防止并发出问题target改为target.long(),防止调用API出现value error
附录
Baseline.py
import glob # 获取文件路径
import numpy as np
import pandas as pd
import nibabel as nib # 处理医学图像数据
from nibabel.viewers import OrthoSlicer3D # 图像可视化
from collections import Counter # 计数统计
# 读取训练集文件路径
train_path = glob.glob('./脑PET图像分析和疾病预测挑战赛公开数据/Train/*/*')
test_path = glob.glob('./脑PET图像分析和疾病预测挑战赛公开数据/Test/*')
# 打乱训练集和测试集的顺序
np.random.shuffle(train_path)
np.random.shuffle(test_path)
# 对PET文件提取特征
def extract_feature(path):
# 加载PET图像数据
img = nib.load(path)
# 获取第一个通道的数据
img = img.dataobj[:, :, :, 0]
# 随机筛选其中的10个通道提取特征
random_img = img[:, :, np.random.choice(range(img.shape[2]), 10)]
# 对图片计算统计值
feat = [
(random_img != 0).sum(), # 非零像素的数量
(random_img == 0).sum(), # 零像素的数量
random_img.mean(), # 平均值
random_img.std(), # 标准差
len(np.where(random_img.mean(0))[0]), # 在列方向上平均值不为零的数量
len(np.where(random_img.mean(1))[0]), # 在行方向上平均值不为零的数量
random_img.mean(0).max(), # 列方向上的最大平均值
random_img.mean(1).max() # 行方向上的最大平均值
]
# 根据路径判断样本类别('NC'表示正常,'MCI'表示异常)
if 'NC' in path:
return feat + ['NC']
else:
return feat + ['MCI']
# 对训练集进行30次特征提取,每次提取后的特征以及类别('NC'表示正常,'MCI'表示异常)被添加到train_feat列表中。
train_feat = []
for _ in range(30):
for path in train_path:
train_feat.append(extract_feature(path))
# 对测试集进行30次特征提取
test_feat = []
for _ in range(30):
for path in test_path:
test_feat.append(extract_feature(path))
# 使用训练集的特征作为输入,训练集的类别作为输出,对逻辑回归模型进行训练。
from sklearn.linear_model import LogisticRegression
m = LogisticRegression(max_iter=1000)
m.fit(
np.array(train_feat)[:, :-1].astype(np.float32), # 特征
np.array(train_feat)[:, -1] # 类别
)
# 对测试集进行预测并进行转置操作,使得每个样本有30次预测结果。
test_pred = m.predict(np.array(test_feat)[:, :-1].astype(np.float32))
test_pred = test_pred.reshape(30, -1).T
# 对每个样本的30次预测结果进行投票,选出最多的类别作为该样本的最终预测类别,存储在test_pred_label列表中。
test_pred_label = [Counter(x).most_common(1)[0][0] for x in test_pred]
# 生成提交结果的DataFrame,其中包括样本ID和预测类别。
submit = pd.DataFrame(
{
'uuid': [int(x.split('/')[-1][:-4]) for x in test_path], # 提取测试集文件名中的ID
'label': test_pred_label # 预测的类别
}
)
# 按照ID对结果排序并保存为CSV文件
submit = submit.sort_values(by='uuid')
submit.to_csv('submit1.csv', index=None)torch_version.py
import os, sys, glob, argparse
import pandas as pd
import numpy as np
from tqdm import tqdm
import cv2
from PIL import Image
from sklearn.model_selection import train_test_split, StratifiedKFold, KFold
import torch
torch.manual_seed(0)
torch.backends.cudnn.deterministic = False
torch.backends.cudnn.benchmark = True
import torchvision.models as models
import torchvision.transforms as transforms
import torchvision.datasets as datasets
import torch.nn as nn
import torch.nn.functional as F
import torch.optim as optim
from torch.autograd import Variable
from torch.utils.data.dataset import Dataset
import nibabel as nib
from nibabel.viewers import OrthoSlicer3D
train_path = glob.glob('./脑PET图像分析和疾病预测挑战赛公开数据/Train/*/*')
test_path = glob.glob('./脑PET图像分析和疾病预测挑战赛公开数据/Test/*')
np.random.shuffle(train_path)
np.random.shuffle(test_path)
DATA_CACHE = {}
class XunFeiDataset(Dataset):
def __init__(self, img_path, transform=None):
self.img_path = img_path
if transform is not None:
self.transform = transform
else:
self.transform = None
def __getitem__(self, index):
if self.img_path[index] in DATA_CACHE:
img = DATA_CACHE[self.img_path[index]]
else:
img = nib.load(self.img_path[index])
img = img.dataobj[:, :, :, 0]
DATA_CACHE[self.img_path[index]] = img
# 随机选择一些通道
idx = np.random.choice(range(img.shape[-1]), 50)
img = img[:, :, idx]
img = img.astype(np.float32)
if self.transform is not None:
img = self.transform(image=img)['image']
img = img.transpose([2, 0, 1])
return img, torch.from_numpy(np.array(int('NC' in self.img_path[index])))
def __len__(self):
return len(self.img_path)
import albumentations as A
train_loader = torch.utils.data.DataLoader(
XunFeiDataset(train_path[:-10],
A.Compose([
A.RandomRotate90(),
A.RandomCrop(120, 120),
A.HorizontalFlip(p=0.5),
A.RandomContrast(p=0.5),
A.RandomBrightnessContrast(p=0.5),
])
), batch_size=2, shuffle=True, num_workers=0, pin_memory=False
)
val_loader = torch.utils.data.DataLoader(
XunFeiDataset(train_path[-10:],
A.Compose([
A.RandomCrop(120, 120),
])
), batch_size=2, shuffle=False, num_workers=0, pin_memory=False
)
test_loader = torch.utils.data.DataLoader(
XunFeiDataset(test_path,
A.Compose([
A.RandomCrop(128, 128),
A.HorizontalFlip(p=0.5),
A.RandomContrast(p=0.5),
])
), batch_size=2, shuffle=False, num_workers=0, pin_memory=False
)
class XunFeiNet(nn.Module):
def __init__(self):
super(XunFeiNet, self).__init__()
model = models.resnet18(True)
model.conv1 = torch.nn.Conv2d(50, 64, kernel_size=(7, 7), stride=(2, 2), padding=(3, 3), bias=False)
model.avgpool = nn.AdaptiveAvgPool2d(1)
model.fc = nn.Linear(512, 2)
self.resnet = model
def forward(self, img):
out = self.resnet(img)
return out
model = XunFeiNet()
model = model.to('cuda')
criterion = nn.CrossEntropyLoss().cuda()
optimizer = torch.optim.AdamW(model.parameters(), 0.001)
def train(train_loader, model, criterion, optimizer):
model.train()
train_loss = 0.0
for i, (input, target) in enumerate(train_loader):
input = input.cuda(non_blocking=True)
target = target.cuda(non_blocking=True)
output = model(input)
loss = criterion(output, target.long())
optimizer.zero_grad()
loss.backward()
optimizer.step()
if i % 20 == 0:
print(loss.item())
train_loss += loss.item()
return train_loss / len(train_loader)
def validate(val_loader, model, criterion):
model.eval()
val_acc = 0.0
with torch.no_grad():
for i, (input, target) in enumerate(val_loader):
input = input.cuda()
target = target.cuda()
# compute output
output = model(input)
loss = criterion(output, target.long())
val_acc += (output.argmax(1) == target).sum().item()
return val_acc / len(val_loader.dataset)
for _ in range(40):
train_loss = train(train_loader, model, criterion, optimizer)
val_acc = validate(val_loader, model, criterion)
train_acc = validate(train_loader, model, criterion)
# print(train_loss, train_acc, val_acc)
print("{:d} epoch".format(_))
print("train loss:{:.5f}".format(train_loss))
print("train_acc:{:.5f}".format(train_acc))
print("val_acc:{:.5f}".format(val_acc))
print("------------------------")
def predict(test_loader, model, criterion):
model.eval()
val_acc = 0.0
test_pred = []
with torch.no_grad():
for i, (input, target) in enumerate(test_loader):
input = input.cuda()
target = target.cuda()
output = model(input)
test_pred.append(output.data.cpu().numpy())
return np.vstack(test_pred)
pred = None
for _ in range(10):
if pred is None:
pred = predict(test_loader, model, criterion)
else:
pred += predict(test_loader, model, criterion)
submit = pd.DataFrame(
{
'uuid': [int(x.split('\\')[-1][:-4]) for x in test_path],
'label': pred.argmax(1)
})
submit['label'] = submit['label'].map({1: 'NC', 0: 'MCI'})
submit = submit.sort_values(by='uuid')
submit.to_csv('submit2.csv', index=None)
更新日志
2023年8月16日:跑通基于Logistic Regression的baseline,分数难看
2023年8月17日:跑通基于CNN的baseline,拿到一个还过得去的分数



